Nat Methods:人类生物分子图谱计划(HuBMAP)新进展:基于多模态数据深度整合,实现3D人类参考图谱多维升级
时间:2025-03-20 12:12:29 热度:37.1℃ 作者:网络
人体生物分子图谱计划(HuBMAP)于2018年启动,旨在构建一个从器官、组织到细胞和生物标志物的健康(“非患病”)人体综合参考模型,使人们理解人体正常生理状态以及疾病发生机制的重要框架。人类参考图谱(HRA)包含一个通用坐标框架(CCF),其有助于整合多模态数据,如3D器官模型、组织学图像和单细胞分析的组学数据。HRA数据包括由人类专家生成的信息(如解剖系统、解剖结构、细胞类型、生物标志物(ASCT+B)表格以及2D、3D参考对象)、映射到HRA的实验数据,以及丰富的图谱数据。
目前,现有图谱使用特定器官的参考系统,不同数据难以在统一框架下进行整合与分析。为推动CCF发展,2020年3月,美国国立卫生研究院(NIH)和人类细胞图谱(HCA)联盟组织成立了HRA工作组。在过去的55个月里,工作组成员共同定义了HRA关键属性及相关标准操作流程(SOP),确定了有意义的数据、代码和工具,为后续多尺度HRA的构建和应用奠定了坚实基础。相关内容以“Human BioMolecular Atlas Program (HuBMAP): 3D Human Reference Atlas construction and usage”为题发表在Nature Methods上。

据悉,HRA工作组与将与跨20多个国际项目合作,通过详细讨论制定了有助于HRA构建、使用和可持续性的三个关键目标,即①将新组织与现有数据进行匹配;②包含能够洞察发生在身体各个层面的变化的功能(如衰老、疾病或其他扰动);③采用能够鼓励协作并指导未来发展的流程,如利用具有模块化、轻量级组件的架构等。此外,对于图谱构建和使用的关键SOP以及HRA术语也达成一致。
在2023年12月发布的第六版HRA v2.0(图1)中,包含4,499种独特解剖结构、1,195种细胞类型和2,089种生物标志物(如基因、蛋白质和脂质),这些数据来自33个ASCT+B表和65个与本体相关联的3D参考对象。
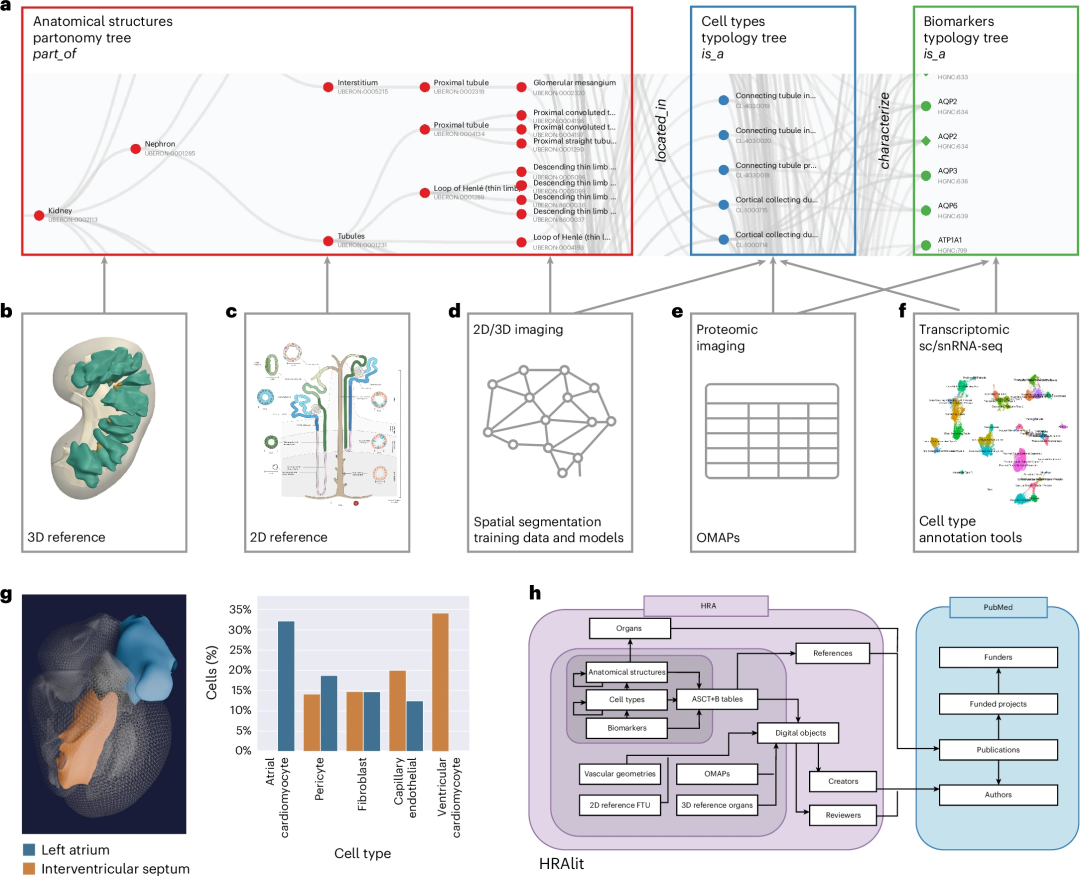
图1. HRA组成部分及关联
为构建复杂而精细的HRA图谱,研究团队运用了多种关键技术方法。首先收集来自不同个体的组织样本,包括多种器官的组织块,并通过三种方式对样本进行数据整合,使用Azimuth进行单细胞注释,通过器官定位抗体panel(OMAP)实现空间蛋白质组定位,注册用户界面(RUI)完成组织样本3D空间注册。
在数据扩展方面,研究团队扩展了Uberon解剖本体(ontologie)和Cell Ontology细胞本体,新增125个解剖术语和141个细胞类型。通过HRAlit数据库将700万篇文献、58万学者信息与图谱数据相关联,HRApop整合了553个组织数据集,用于计算具有3D参考对象的40个解剖结构的细胞类型。
研究团队还开发了灵活的混合云架构,以支持数据的管理、分析和访问。该架构整合了50多个开源算法,实现了数据的半自动提取、自动分析注释、可查找性、可访问性、互操作性、分析支持和可持续性。在这个架构下,数据和代码通过HuBMAP数据门户和HRA门户提供服务,方便研究人员使用。
此外,研究团队开发了统一的数据处理流程,涵盖多种数据类型的处理。例如,针对单细胞(sc)/ 单细胞核(sn)RNA 测序数据,基于Salmon准映射方法构建了处理流程,并使用Scanpy和scVelo进行下游分析和RNA速度计算。对于成像数据,使用端到端的分析方法,包括图像校正、细胞和细胞核分割等步骤,并通过空间过程和关系建模(SPRM)计算相关指标。
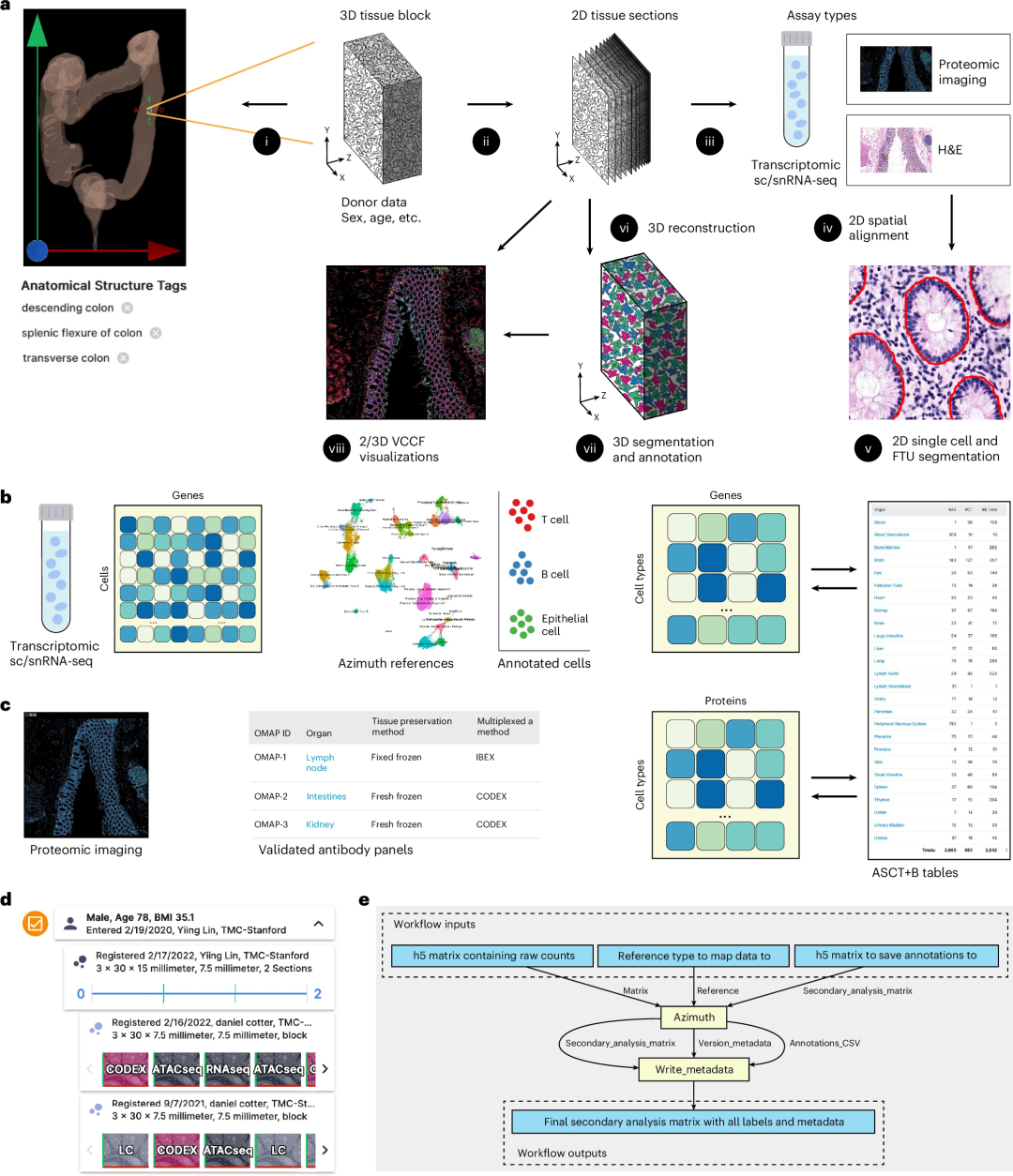
图2. 将实验数据映射到HRA
为方便用户访问和分析HRA数据,研究团队开发了多种用户界面、工具及接口。HuBMAP门户(https://hubmapconsortium.org)介绍了项目目标,并提供了实验数据、图谱数据、工具等资源导航;HuBMAP数据门户(https://portal.hubmapconsortium.org)提供了实验数据集,并支持处理、搜索、筛选和可视化数据;HRA门户(https://humanatlas.io)提供对图谱数据、代码和程序等开放访问,支持使用和下载。
ASCT+B Reporter用于ASCT+B表和OMAPs的编写和审核;Azimuth用于自动化处理和分析sc/snRNA-seq和ATAC-seq数据。特别地,Vitessce用于可视化探索实验数据、Azimuth参考、HRA 分割注释或细胞间距离分布可视化。注册用户界面(RUI)用于将人体组织块注册到三维CCF中;探索用户界面(EUI)支持在全身器官、组织和细胞层面浏览组织样本和元数据。交互式功能组织单元(FTU)页面用于在2D空间背景下探索细胞类型和生物标志物表达矩阵。
研究团队计划每6个月发布一次新的HRA版本,通过HRA门户发布更新的数据对象(DOs)、用户界面和API。每次发布都会扩充现有本体(ontologie),改进HRA数据结构和算法,以更好地满足国际人类地图绘制界的需求。HRA的核心本体(样本、生物结构和空间本体)将遵循可查找、可访问、可互作、可重用(FAIR)原则、以版本化的链接开放数据(LOD)形式共享,部分数据以关系数据库和CSV文件形式提供。同时,HRA的来源图会记录所有DOs和代码版本,以保证数据的可追溯性和可重复性。
最后,研究人员展示了HRA的应用价值。在肺组织研究中,通过HRA比较健康和患病肺组织中细胞与血管的距离分布,发现患病肺组织中免疫细胞聚集在血管附近,导致气体交换膜增厚、影响肺功能,为理解肺部疾病的病理生理机制和靶向治疗提供了依据。在肠道组织研究中,使用HRA能够在不同尺度上分析和展示FTU的结构和功能,有助于揭示肠道组织的奥秘。
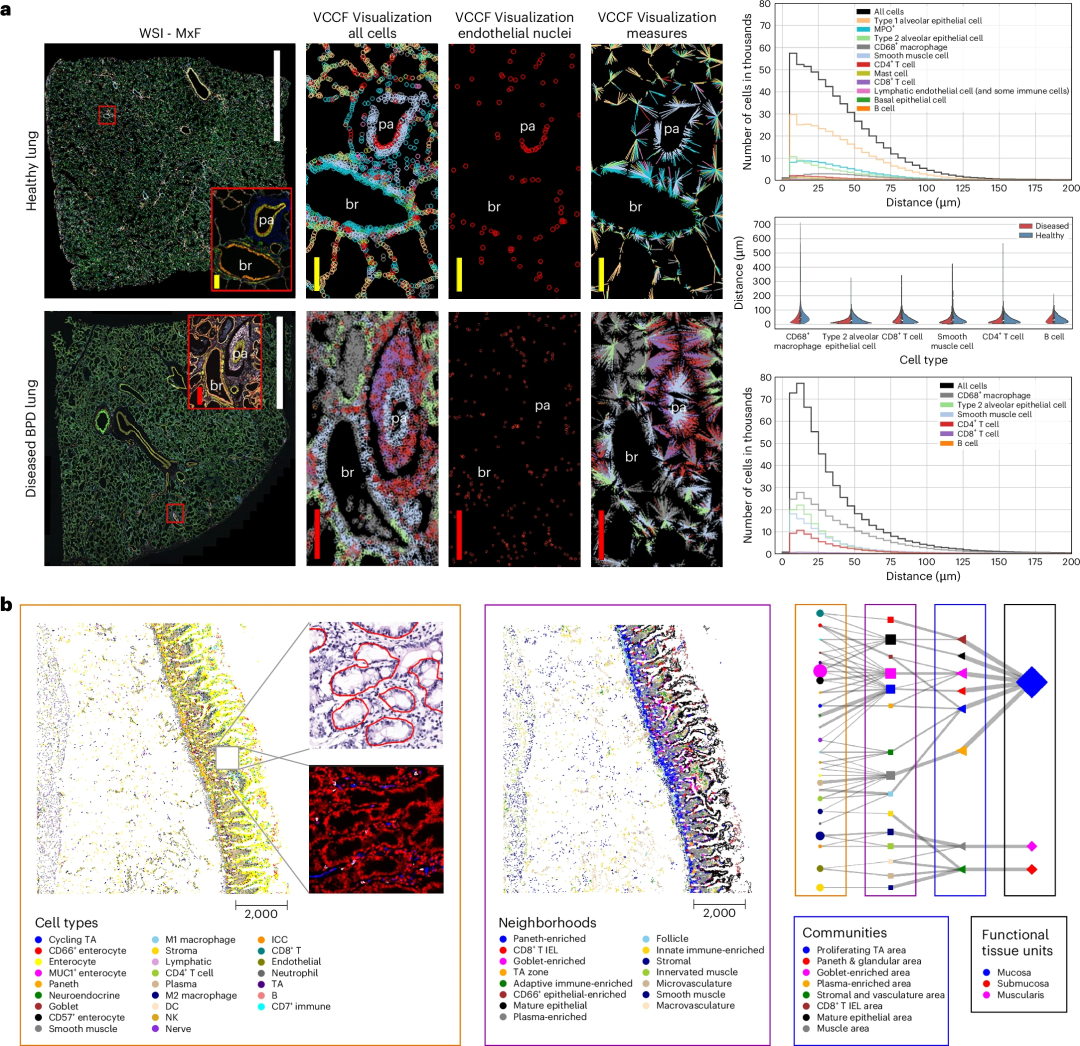
图3. HRA的应用情况
目前,HRA知识图谱(KG)和API在HuBMAP、SenNet、GUDMAP、GTEx数据门户和CZ CellGuide中驱动不同的2D和3D用户界面。研究团队也正在开发额外的轻量级web组件,以便人们访问HRA数据和在其他网站中提供HRA功能。
参考文献:
Börner, K., Blood, P.D., Silverstein, J.C. et al. Human BioMolecular Atlas Program (HuBMAP): 3D Human Reference Atlas construction and usage. Nat Methods (2025). https://doi.org/10.1038/s41592-024-02563-5


