Circ Res 北京大学郭宇轩联合北京安贞医院高霏课题组发表单AAV递送的心肌特异性单碱基编辑技术及其应用
时间:2025-01-19 12:09:11 热度:37.1℃ 作者:网络
腺嘌呤碱基编辑器(ABE)是在CRISPR/Cas9基础上衍生的基因编辑工具,能够实现基因组中A•T到G•C碱基的转换。ABE可以纠正基因突变或消除翻译后修饰,具有心脏基因治疗的潜力1。但是受到序列环境和染色质可及性等因素影响,ABE的编辑效率是高度可变的并且难以预测,这使得在指定靶碱基的情况下,ABE基因编辑的成功率有限。相比之下,利用ABE进行基因沉默成功率更高,因为这种策略可以靶向基因的起始密码子和剪接位点设计多条sgRNA并进行筛选。目前唯一在临床上得到验证的ABE心血管基因编辑药物即采用基因沉默的策略(NCT05398029)。
ABE在心脏中应用的另一大限制在于其较大的体积。受到AAV载荷限制,双AAV载体是目前心脏ABE递送的主要工具2,这限制了AAV基因治疗的递送效率、安全性和成本。基于小型化Cas9变体的ABE可以被单AAV装载,但这些小型化的ABE能否高效地应用于心脏尚不清楚。单AAV载体在心脏基因编辑中是否确实优于双AAV缺乏实验证据。
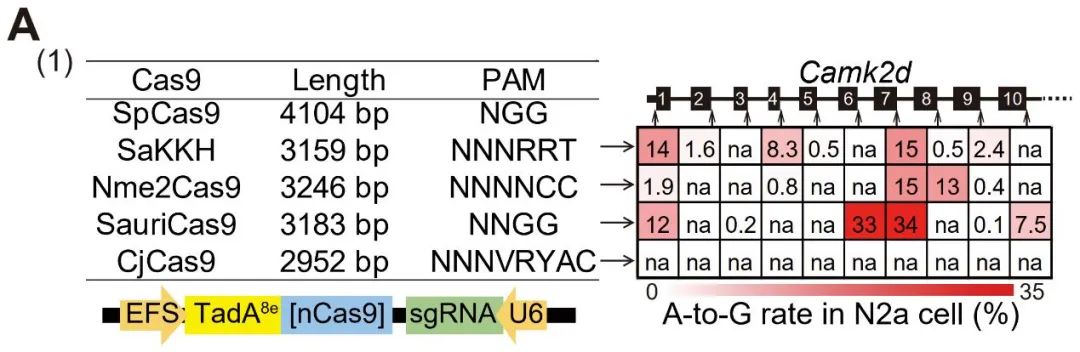
为了解决这些问题,北京大学郭宇轩研究员联合安贞医院高霏教授在Circulation Research发表题为“ABE-Mediated Cardiac Gene Silencing via Single AAVs Requires DNA Accessibility”的文章,开发了单个AAV递送的心肌特异性的单碱基编辑技术,强调其在心脏基因沉默中的应用,以及可能受心肌特异性染色质可及性的调节3。作者首先采用了SaKKH、Nme2Cas9、SauriCas9和CjCas9等4种小型Cas9构建的ABE4,选择心脏疾病的广谱治疗靶点CaMKIIδ的编码基因Camk2d作为靶基因,靶向Camk2d的起始密码子和5’剪接位点(5’SS)设计了18条sgRNA。在N2a细胞中筛选确定了SauriABE的具有高编辑活性的sgRNA1、6、7 (图A(1))。
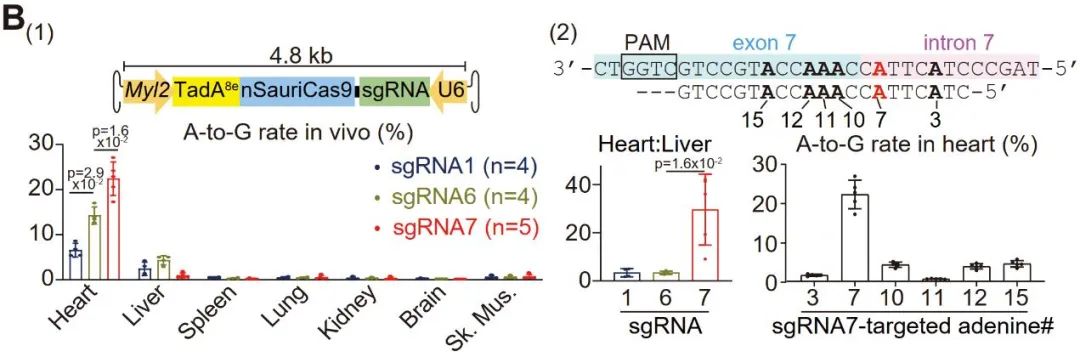
在体内实验中,为了尽可能节省AAV中的空间,作者使用了较小的心脏特异性Myl2启动子(<300bp)5。对出生后1天(P1)的小鼠皮下注射了携带ABE基因组的AAV9 (1x10^11vg),并在P7收集心室组织用于扩增子测序。结果表明,sgRNA7表现出最好的编辑效率和心脏特异性,并且优先编辑5’SS腺嘌呤而不是旁观者腺嘌呤(图B)。
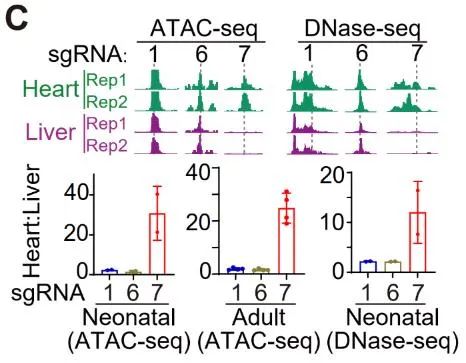
接下来,作者分析了多个公共数据库中的ATAC-seq和DNase-seq数据,比较3条sgRNA靶向位置的染色质开放程度。结果表明,sgRNA7靶向的基因位置在心脏中是开放的,而肝脏中不开放,与sgRNA7独特的心肌特异性活性一致。这表明sgRNA的器官选择性与组织特异性染色质开放有关(图C)。
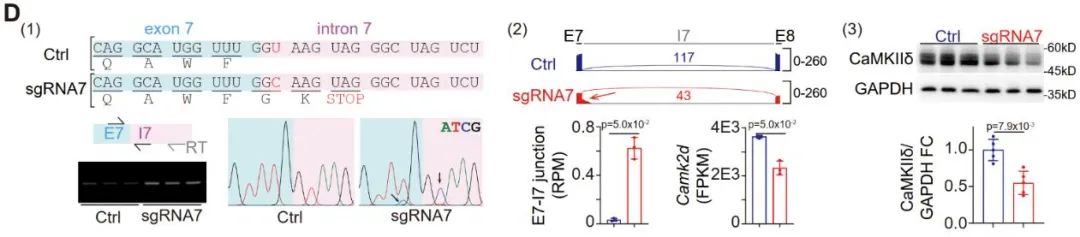
作者进行的逆转录-PCR (RT-PCR)结果表明,sgRNA7处理组Camk2d mRNA表现出内含子7保留增加,Sanger测序在RNA层面证实了对内含子7的5’SS编辑(图D(1))。RNA-seq进一步验证了内含子7保留增加,并且Camk2d的总mRNA显著下降,这可能是无义突变介导的mRNA衰变导致的(图D(2))。与RNA变化一致,Western blot也验证了CaMKIIδ蛋白降低(图D(3))。
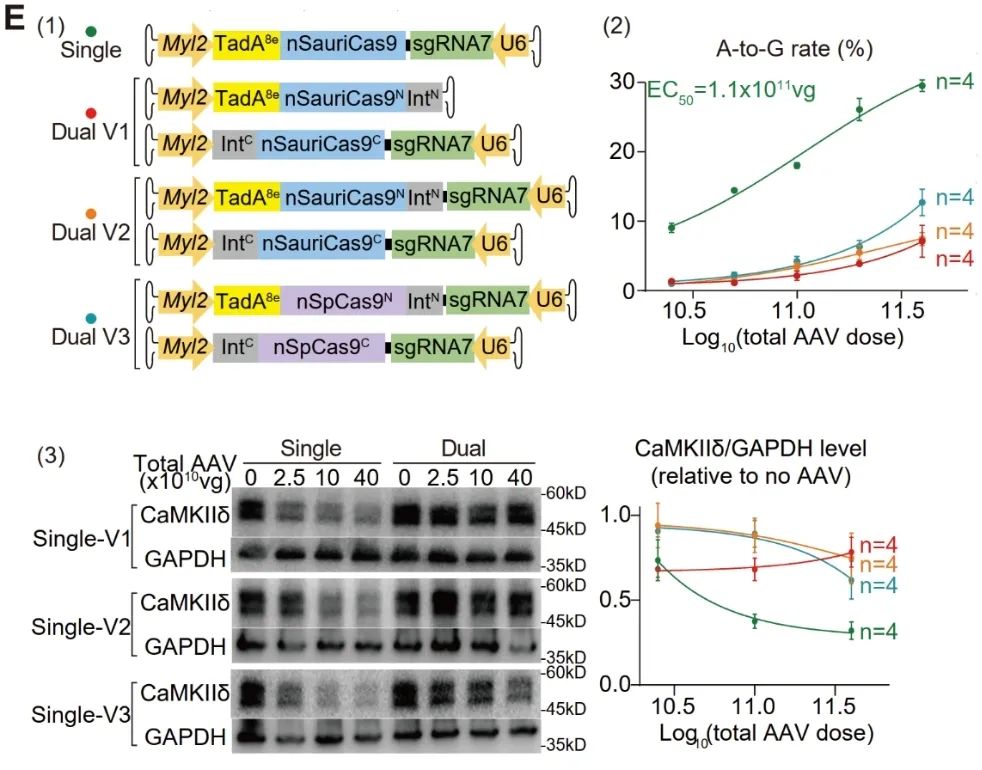
接下来为了对比单AAV与双AAV系统,作者构建了三种双AAV载体作为对照,分别是:携带单个U6-sgRNA7拷贝(V1)和携带两个U6-sgRNA拷贝(V2)的双AAV-SauriABE载体,以及携带两个U6-sgRNA拷贝的SpCas9-ABE (图E(1))。作者进行了一系列的AAV剂量梯度实验,并分析“量-效”关系。结果表明在所有剂量下,单AAV的编辑效率和蛋白敲除效果都优于双AAV (图E(2-3))。
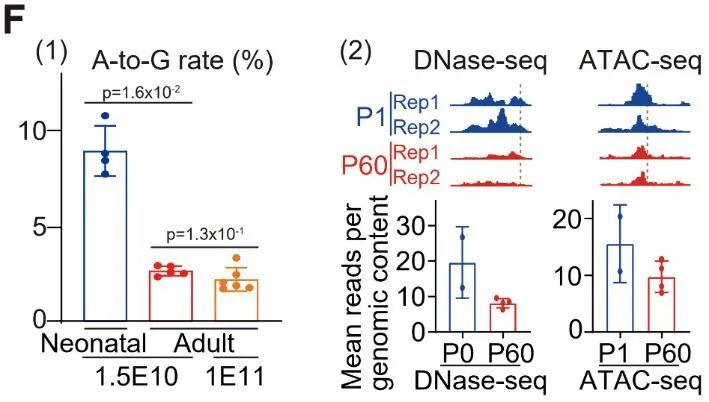
最后作者对比了新生和成年小鼠的编辑效率,发现新生阶段的编辑效率高于成年阶段(图F(1)),这与成年小鼠心脏中较低的染色质可及性相关(图F(2)),并且无法通过增加AAV剂量来克服。
总之,本研究表明,利用多样化的Cas9同源物可以通过筛选大量sgRNA,促进单AAV递送的心脏碱基编辑并进行基因沉默,提高ABE在心脏中应用的成功率。本研究的结果提示染色质可及性等表观遗传特征可能会对基因编辑结局产生器官特异性和年龄依赖性的影响,在未来设计心脏基因编辑策略时应系统评估。同时,本研究提供了实验证据证明在心脏基因编辑中单AAV相对于双AAV载体有优势。
北京大学基础医学院2021级博士生刘占钊和杨璐梓是本研究的共同第一作者,2020级基础医学八年制本科生杨喻涵在生物信息学分析中提供了重要帮助。北京大学郭宇轩研究员和北京安贞医院高霏教授是本研究的通讯作者。本工作获得了北京大学赵东宇研究员和北京协和医院胡晓敏研究员的大力支持与合作。本研究获得国家自然科学基金、国家重点研发计划和北京安贞医院高水平研究基金的资助。
参考文献:
1. Reichart D, Newby GA, Wakimoto H, Lun M, Gorham JM, Curran JJ, Raguram A, DeLaughter DM, Conner DA, Marsiglia JDC, et al. Efficient in vivo genome editing prevents hypertrophic cardiomyopathy in mice. Nat Med. 2023;29:412-421. doi: 10.1038/s41591-022-02190-7
2. Lebek S, Chemello F, Caravia XM, Tan W, Li H, Chen K, Xu L, Liu N, Bassel-Duby R, Olson EN. Ablation of CaMKIIdelta oxidation by CRISPR-Cas9 base editing as a therapy for cardiac disease. Science. 2023;379:179-185. doi: 10.1126/science.ade1105
3. Yuan T, Wu L, Li S, Zheng J, Li N, Xiao X, Zhang H, Fei T, Xie L, Zuo Z, et al. Deep learning models incorporating endogenous factors beyond DNA sequences improve the prediction accuracy of base editing outcomes. Cell Discov. 2024;10:20. doi: 10.1038/s41421-023-00624-1
4. Davis JR, Wang X, Witte IP, Huang TP, Levy JM, Raguram A, Banskota S, Seidah NG, Musunuru K, Liu DR. Efficient in vivo base editing via single adeno-associated viruses with size-optimized genomes encoding compact adenine base editors. Nat Biomed Eng. 2022;6:1272-1283. doi: 10.1038/s41551-022-00911-4
5. Bilal AS, Blackwood EA, Thuerauf DJ, Glembotski CC. Small Nppa and Myl2 Promoters Are Sufficient to Maintain Chamber-specific Expression on an AAV9 Platform. Faseb J. 2020;34. doi: 10.1096/fasebj.2020.34.s1.07372


