【佳作推荐】佛罗里达国际大学Narasimhan小组NMI论文:使用Transformer网络评估蛋白结合界面
时间:2023-11-06 22:45:04 热度:37.1℃ 作者:网络

随着生物实验积累和人工智能蛋白结构预测模型的发展,蛋白质三维结构的获取更为方便快捷,这为基于结构的大分子药物发现提供了条件。蛋白质之间的相互作用是多种功能的分子机器的结构基础,调控和主导许多生理活动过程。然而,蛋白质结合界面的评估和精确打分仍然是一个挑战,主流的分子表面相互作用指纹 (MaSIF) 模型采用界面patch特征来预测蛋白质相互作用,此方法仅从蛋白单体分别采样,忽略了蛋白复合物界面的相互作用特征。
针对此现状,佛罗里达国际大学Narasimh小组对MaSIF工程架构加以改进,提出了一个深度学习模型,即蛋白界面打分Transformer模型(Protein Interface Scoring with Transformer Network ,PIsToN),用于准确高效地评估蛋白-蛋白对接。近日,该项研究工作发表在Nature Machine Intelligence上(Nat. Mach. Intell. 2023,5, 1042-1053)【1】。
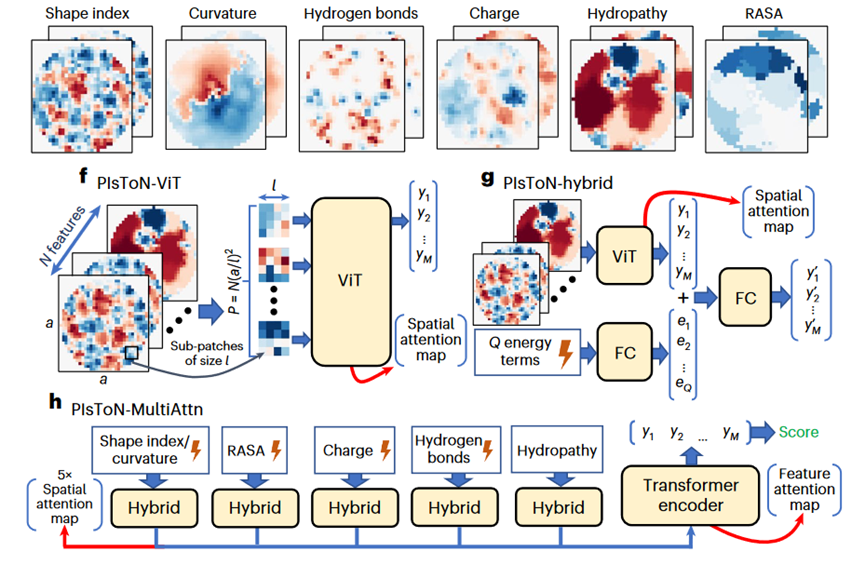
图1: PIsToN网络架构示意图
PIsToN将蛋白界面特征(几何形状、电荷、亲水性和相对溶剂可及表面等)分布图降维至2D图输入Vision Transformer (ViT)层(图1f),像素强度代表特征值并进行了若干增强,输出特征和FireDock计算的经验能量项拼合输入全连接层FC(图1g),使得网络能够感知详细的界面特征分布和全局近似能量。 PIsToN将为界面空间和电荷、亲水性等各理化特征分配注意点,网络的输出通过Transformer编码器进行组合,以产生最终的分数。(图1h)
PIsToN使用多注意力网络,将每类界面特征通过独立的网络学习后再进行组合,能够在空间注意力图(图2c)中识别阳性(上)与阴性(下)样本的对接界面(图2d),具有更强的可解释性。
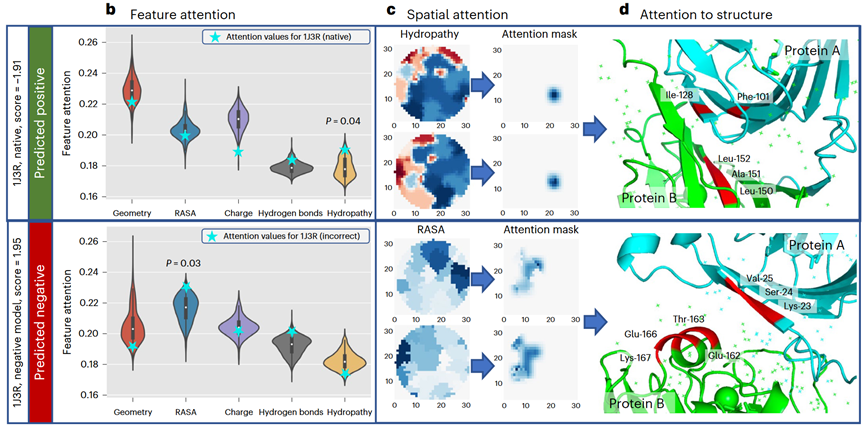
图2: PIsToN在界面特征的预测中具备可解释性
PIsToN训练集包含由与MaSIF相同的5123个天然蛋白质复合,使用HADDOCK对接生成了17291个阳性样本和452031个阴性样本,随后根据CAPRI (Critical Assessment of Predicted Interactions)【2】分配标签。PIsToN在MaSIF-test, CAPRI-score 和PDB-2023三个测试集上进行了蛋白-蛋白结合的分类和排序任务测试,在AUCROC和精度等指标上优于现有RosettaDock、dMaSIF等主流蛋白质界面评估软件。PIsToN使用对比学习以获得更好的排序性能,还能够通过在实验结合亲合力的蛋白质复合物数据集(例如 PDBbind)上进行额外训练来增强其对蛋白界面多结合位点的选择性和结合构象的排序能力。
对于给定一个真实的蛋白-蛋白复合物体系,PIsToN的Top10准确率为69%,远高于第二名HADDock的46%。另外,PIsToN流程中的Patch 是独立预先计算的,这使得其计算效率高于其他方法,大量节约在筛选成千上万个候选大分子高通量虚拟筛选任务中耗费的计算资源。
小结:本文介绍了用于评估蛋白-蛋白作用界面的深度学习模型PIsToN。PIsToN在分类和排序任务中展现了优于其他模型的蛋白界面评估能力,其特征编码方式具有可解释性,能够加快蛋白-蛋白互作虚拟筛选速度。值得一提的是,PIsToN在许多对接任务中具有应用潜力,可用于扫描大量的抗体-抗原的交叉反应;甚至能够拓展应用于蛋白与小分子配体相互作用的虚拟筛选。PIsToN对MaSIF特征工程优化带来的巨大性能和效率的提升,表明了工程架构对于高通量应用程序的重要性。
特别提醒:该工作中描述的网络模型的源代码可从GitHub平台获取(https://github.com/stebliankin/piston)。
参考文献
【1】Evaluating protein binding interfaces with transformer networks. Stebliankin, V., Shirali, A., Baral, P. et al. Nat Mach Intell 5, 1042–1053 (2023).
【2】CAPRI: a Critical Assessment of PRedicted Interactions. Janin, J. et al. Proteins, 52(1), 2–9. (2003).


